|
(厦门大学自动化系模式识别与智能系统研究所,福建 厦门 361005)陈福振,陈光磊

陈福振(1985-)男,福建莆田人,厦门大学自动化系硕士研究生,主要从事模式识别与智能系统方面的研究。
基金项目:福建省自然科学基金(2009J05153)
摘要:软件内嵌探针测试是在探针函数模板的框架内编写被测对象,并对被测对象进行面向对象软件的常规测试,在观察预期结果与实际结果是否一致的同时,还要察看探针函数输出的信息,以确定对象的状态是否正确。本论文研究的重点在于探针函数的构成、探针函数的实现及内嵌探针测试方法的应用研究,意在构筑一个基本的应用框架,提供一种实用的测试方法。
关键词:软件内嵌探针;探针函数;测试
Abstract: his article studies the automatic extraction of gene chip image information. In view of the difficulties in automatic processing, such as the huge number of the DNA spots, the low resolution ratio and the anomalous contour, this article proposes an automatic image division algorithm provided with high efficiency in spot localization and compact spot outline edge. Through the effective selection of structure operator, the nimble application multi-layer mathematics morphology's transformation, and the automatic binaryzation using the difference and standard deviation, this algorithm can automatically read genome image information. Through many experiments of reading genome image information, we also show that this algorithm obtain a compact image segmentation and high precision, in which its relative error with commercial software is less than 5%.
Key words: DNA chip; Mathematics morphology; Image segmentation
1 引言
微阵列芯片,又称为基因芯片,它将大量DNA探针分子或经纯化的蛋白分子通过点样固化于玻片上,构成高密度的分子阵列,再利用特定的仪器对样本进行扫描成像[1]。基因芯片的每个样点表达了一定的生物信息,微阵列芯片技术可对成千上万的基因进行并行分析,该技术广泛应用在疾病诊断、药物筛选及基因测序等生命科学领域。完整的微阵列生物芯片分析过程包括样本采集、芯片制备、扫描成像、图像处理和数据分析等几个部分,其中图像处理的目的是准确地提取基因表达的荧光信号强度值,为后续的分析提供数据基础,图像处理提取的信息正确与否,直接决定后续数据分析的意义。
传统的基因芯片图像处理主要包括基因点定位和信息提取两个步骤,基因点定位分为网格定位和斑点分割,用于确定各基因点的位置,信息提取是在分割出基因点与背景区域后对该点的亮度表达值进行计算。
由于原始图像中的荧光斑点以矩阵形式排列,故传统的基因点定位方法,大多是先做倾斜校正,然后定位斑点的矩形区域(网格定位),再分割出更紧凑的斑点边缘区域(斑点分 割)。在网格定位方面,主要有基于投影的网格定位[2,3],该方法运算速度快,但容易受噪声影响,导致定位失败。基于爬山法的网格定位[4],通过寻找局部最优方式逐步迭代来进行网格定位,初始点的设定将直接影响计算结果。基于马尔可夫随机场的网格定位方法[5]利用不同应用限制和启发式规则进行网格定位,需要给定每个网格内基因的行列数和网格行列数。基于遗传算法的网格定位[6]通过计算间距和初始行列位置进行网格定位,需要设定较多参数,运算耗时。
网格定位后,对斑点进行分割的方法有固定圆形法[7,8],采用固定圆形方式进行邻域搜索,不适用于其他形状的基因点分割;自适应圆形法[9]利用大小可变的圆形进行邻域搜索分割,但受点样探针形状及点样过程玻片滑动等因素的影响,难以找到非绝对圆形的基因点;自适应形状法以分水岭算法或种子区域生长算法为基础,可进行可变形状的基因点分割,但该方法种子选取困难、标记迭代次数多、复杂度高;肖松山对圆形定位处理过程进行了分类讨论,将样点分成“圆域、圆环和不规则区域三类”,并提出相应的“模板匹配法,霍夫变换法和形心法”等三种自动识别算法,但该文献还是以圆形作为基本模板进行处理,变换所用的算法复杂度较大,实验结果并没有与商业软件进行对比或将其与最终的表达数据进行分析;直方图分割法利用一个比任何靶点都大的目标模板对待分割区域进行套框操作,对每个套框中的像素点进行直方图分析,按照预定义的光密度比率分割出信号和背景,该方法的优点在于算法简便、速度快,缺点是目标模板的大小对分割结果有较大影响,造成稳定性差、重复性不好,此外,该方法的分割结果往往会得到一个非连通的前景区域,与点样的实际结果不符合,对于弱信号靶点的分割,直方图分割法的缺点表现得更为明显,应用仍较少。
综上所述,现有的基因芯片图像处理方法普遍存在如下几个问题:(1)自动化程度低,每种方法都涉及到多个参数的预设,有些参数是随机的,有些参数是与图像相关的;(2)靶点分割不准确,由于传统方法采用的是网格定位后进行基因点分割,网格定位的误差将直接向后累加,导致基因点分割失败。基于此,考虑到数学形态学的基本思想是用具有一定形态的结构元素去量度和提取图像中的对应形状,以达到对图像分析和识别的目的。因此根据基因斑点自身不规则的形态特征,本文提出了基于数学形态学的基因芯片图像分割方法,在闭运算对斑点周围进行噪声清除的基础上,结合开运算对斑点的形态进行修复弥补,最终紧凑地进行斑点分割。本文的方法实现了基因芯片图像的形态学分析和自适应二值化,大大提高了图像分析和处理的速度与精度。
2 图像的自适应提取
2.1 流程改进
基因芯片图像处理的一种常用方法是投影法,处理步骤如表1,由于受噪声等影响,很容易产生矩形框误定位,从而导致基因点分割错误,如图1所示。大块高亮噪声会导致矩形框定位冗余,此外,矩形框定位有时会压在基因点上,如果再在矩形框内进行圆形定位,可能会导致基因点不完整,或完全找不到基因点。
表1 处理步骤对比
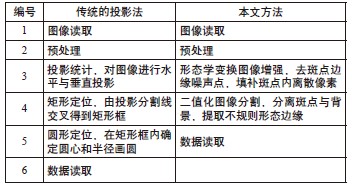


图1 投影法误定位情况
为避免投影网格定位产生的误定位问题,本文提出一种新的基因点分割方法,如表1所示。为使离散的斑点进一步丰满起来,方便精确地刻画其边缘,引入了数学形态学方法,在对其增强的基础上,利用差分运算获取分割阈值进行斑点与背景的分离,从而确定基因点进行数据读取。
2.2 自适应图像增强
传统的图像增强方法一般采用灰度映射法,需要设定较多阀值来界定输入输出的灰度范围,并需要反复实验提取经验值。为提取斑点自身的不规则形态边缘,并去除斑点边缘离散噪声的影响,利用形态学的开闭运算进行处理。应用闭运算来填充斑点内细小空洞、连接邻近像素点、平滑其边界的同时并不明显改变其面积。再应用开运算消除离散噪声像素,在纤细点处分离斑点与噪声。此外,由于数学形态学的开闭运算具有极值滤波功能,开运算增大了谷值,扩展了峰顶,是极大值滤波;闭运算减少了峰值,加宽了谷域,是极小值滤波。基于此,本文提出一种自适应图像增强算法如下:
(1)用原图像减去开运算后的图像获得峰值, 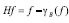 ,其中f 为原始图像,r 代表开运算, B为结构算子 ,其中f 为原始图像,r 代表开运算, B为结构算子
(2)用闭运算后的图像减去原图像获得谷值,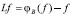
(3)增强图像为 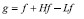
应用数学形态学进行图像增强处理的关键在于结构算子的选择,由于DNA芯片与经过荧光标记的样品杂交后,产生的荧光图像中包含一系列类圆形的斑点,故选择圆形结构算子,其直径为基因点直径。增强效果对比情况如图2所示。

(a) 原图部分区域 (b)传统图像增强效果 (c)本文方法增强后的效果
2.3 自适应二值化图像分割
形态学增强后的斑点边缘基本清晰可见,可以较容易地提取出边缘。但为了后续数据进行斑点连通域索引,需要对图像进行二值化处理。把斑点区域像素位置记为1,把背景像素位置记为0。这样可以省去很多边缘提取的复杂计算,又为后续图像分割减少计算量。
由形态学增强后的图像直方图可见,如图3a所示,虽然没有明显的双峰效果,基本是呈一个下降的趋势,且有从陡到平的过渡过程,很难用常规阈值分割方法进行处理。 从直方图中图像背景的大范围分布可以看出其对应灰度值的集中性,此外,在坡度放缓之后有一段较为平整的灰度带,表明各个斑点虽然内部灰度值区别较大,但总要经过一个灰度值从背景与斑点内部的过渡。 因此,本文提出利用灰度的一阶差分来观察灰度值之间的变化趋势,从而找出分割阈值,图3b给出了灰度的一阶差分效果。

(a) 区块对应直方图
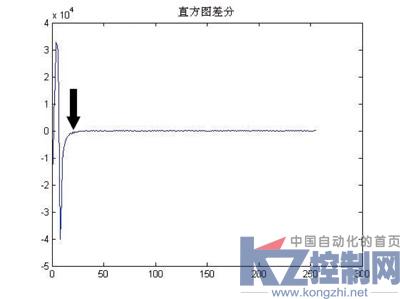
(b) 区块对应直方图一次差分效果,箭头为阀值点
(b)对区块直方图一次差分效果,箭头为阀值点图3 对图1所在22×22整个区块进行形态学变换后的分析结果利用标准差来评估差分曲线可能的变化或波动程度。标准差越大,差分的范围就越广,差分的波动就越大。

其中, n 为图像灰度直方图差分的最大范围, xi为当前点的差分值,x'为平均值。
但本文并不关心标准差本身的大小,而是关心差分序列中的哪个灰度值开始接近或等于标准差,即能体现背景与斑点的最大差别,并把此灰度值作为二值化的阈值。根据实际的差分曲线波动由大到小,灰度由背景向斑点过渡的特点,只从小灰度开始搜索,这时的差分值由比标准差大到比标准差小变化,找到第一次差分序列中绝对值开始接近(实际程序取开始小于)标准差的灰度值,即为二值化阀值。具体如下:
(1)为方便计算,首先将16位灰度图转换为8位灰度图g' =g / 256;
(2)获取增强后图像直方图dh(g');
(3)计算直方图的一次差分序列sd;
(4)计算差分曲线的标准差;
(5)判断灰度级下的绝对差分值与标准差的关系,如果小于标准差则表示第一次接近标准差,转(6),否则重复步骤;
(6)获取分割阈值。二值化后的基因芯片图像如图4a所示。
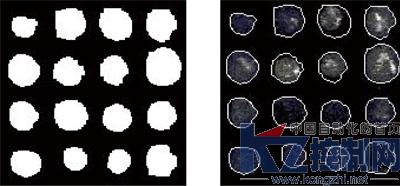
(a) 对图1进行二值化的后效果 (b) 形态学边缘提取法
图4 图像提取
2.4 数据读取
对二值化后的图像中进行0,1索引,对于每个连通域,即每个斑点,逢1即以当前坐标为准返回原图读取像素值,得到如图4b所示的边缘提取效果。在背景灰度扣除上,我们利用形态学外围像素的中值作为背景值,该值能较为准确地说明其周边背景的分布情况,从后续的实验分析中我们可以看到此方法确实效果更好。
3 实验分析
为检验本文算法的效率与准确性,我们针对美国Baylor医学院提供的10G多的图像数据进行对比实验。计算机仿真实验在Matlab7下完成。
3.1 斑点边缘分割形态对比
图5为采用投影分割圆形定位法(如著名的GenePix Pro与ScanAlyze软件)与本文的形态学边缘提取法定位各斑点的对比效果图;为使对比清晰,底层为图像增强后的图。

(a)投影分割在图像增强后的效果 (b) 图形定位在图像增强后的效果 (c)形态学边缘在图像增强后的效果
图5 边缘提取的效果对比图
从图5可看出,本文的方法(图5c)可以更为完整并精确地定位斑点。本文的方法之所以可以更为完整和精确地定位斑点,是因为:(1)本文的方法由于没有用到倾斜校正与坐标投影,不存在倾斜校正的样本选取、算法复杂度的问题和投影分割阀值设定的问题。(2)本文的方法直接通过形态学的方法圈定其紧凑的类椭圆的紧凑轮廓,而非投影分割法中带有噪声的矩形框(图5a)和需经过圆心定位,半径取值后再绘制的圆框(图5b);从图5中的三组对比可以清晰地看到一些离散的噪声点被形态学处理后的边缘轮廓排除在外,而前两组则无法将背景像素完全排除在外。(3)本文的方法二值化处理后,只要跟踪图像中非0像素,再对应到原图,即可读取原像素值;这样既降低了原来需要边缘提取的算法复杂度,又对前一步处理留下的噪声进行进一步的清除。
3.2 数据提取对比
图6是本文方法对cy5与cy3的散点图,图7是本文的方法与业界公认的权威分析软件Bluefuse数据读取的相对误差图。
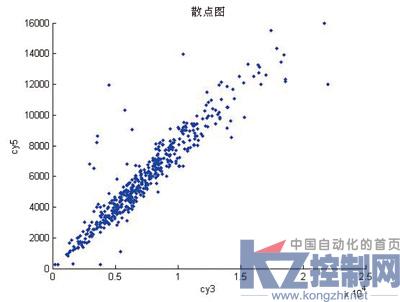
图6 cy5与cy3图像数据散点图
从图6可看出,散点图中呈现趋势统一的拟合直线,说明本文提取的数据是较为精确的。另外,数据点分布在散点图零点区域的多少可体现基因数据提取的完整度。从图6的零点区域散点稀少也可以看出本文提取数据的完整度较高。从图7可以明显看到本文方法与Bluefuse软件的相对误差基本保持在0.05以内,并集中在0.02左右,较为充分地说明了本文方法算法的准确性。
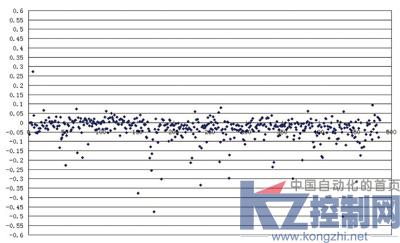
图7 Bluefuse软件对比本文算法的相对误差图
4 结束语
本文利用数学形态学和自适应二值化方法,在实现基因芯片图像数据自动提取的同时,也获得了可竞争的读取精度。与其他软件产品,如GenePix Pro与ScanAlyze 的手动网格定位,手动调节半径等过程对比,本文的方法可以更容易更快捷地得到DNA芯片的图像分割与斑点数据信息。实验证明该算法大大提高检测的效率和准确度,为后续生物信息学数据挖掘,模式识别分析提供可靠的数据支持。
参考文献:
[1] 刘长春, 文孟良. 生物芯片[J]. 分析仪器, 2001(3): 37-39.
[2] Luis Rueda and Bidya Vidyadharan. A hill-climbing approach for automatic gridding of cDNA microarray images[J]. IEEE Transactions on Computational Biology and Bioinformatics, 3(1),2006:72-83
[3] Zacharia, E.,Maroulis, D.,An Original Genetic Approach to the Fully Automatic Gridding of Microarray Images[J]. IEEE Transaction on Medical Imaging,2008, 27(6):805-813.
[4] Antonio P.G. Damiance Jr., Liang Zhao, Andre C.P.L.F. Carvalho.A dynamical model with adaptive pixel moving for microarray images segmentation[J] . RealTimeImg(10) ,2004, (4): 189–195.
[5] Glasbey C A ,Ghazal P. Combinatorial Image Analysis of DNA Microarray Features. Bioinformatics, 2003,19 (2) :1942203.
[6] 肖松山. 生物芯片图像自动识别算法研究[J]. 生命科学仪器. 2003.
[7] 马驰, 张红云, 苗夺谦, 张学东. 改进的多阈值动态二值化算法[J]. 计算机工程. 2006. 3.
[8] 崔屹. 图像处理与分析——数学形态学方法及应用[M]. 北京: 科学出版社. 2000. 4. 67-77.
摘自《自动化博览》2010年第十一期
|




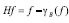 ,其中f 为原始图像,r 代表开运算, B为结构算子
,其中f 为原始图像,r 代表开运算, B为结构算子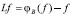
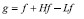



















.jpg)



